Anwendungen zur Medizinischen Bildverarbeitung
3D Ultrasound bei Prostatabiopsien
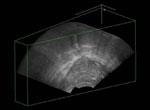 |
Die Anwendung "3D Ultraschall" umfasst die Auswertung und die
Darstellung von Biopsieergebnissen bei der männlichen Prostata in
einem 3D visualisiertem Bild. Das bei der Untersuchung entstehende
und dafür nötige Bildmaterial sind sowohl klassische 2D
Ultraschallbilder und -filme als auch 3D Ultraschallbilder. Wie bei
allen film- und dreidimensionalen Bildgebungsverfahren ist das
entstehende Datenmaterial sehr groß. Als Folge ist die Arbeit mit
diesen Bildern aufwändig, zeitraubend und vielschichtig. |
Zur Anwendung im MediGRID Applikationsportal
Virtuelle Gefäßchirurgie
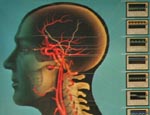 |
Die Strömungssimulation in Blutgefäßen dient der 3D
Darstellung von Blutströmung in Gefäßen und deren Veränderung.
Die Geometrie der Gefäße wird durch unterschiedliche
Aufnahmemodalitäten erfasst. Anschließend wird die Blutströmung
und die resultierende Belastung der Gefäßwände berechnet. Eine
zu hohe Gefäßbelastung bildet die Gefahr einer Blutung, die bei
Hirngefäßen zum Schlaganfall führt.
Die Strömungssimulation liefert eine animierte 3D Darstellung der
Gefäße mit Färbung der Gefäßwände in Abhängigkeit von der
Druckbelastung während einer Pulsperiode. Der selbstentwickelte
Algorithmus liefert im Vergleich zu existierenden Verfahren
schnelle, einfache, zuverlässig aufbereitete Resultate und
überwindet Schwächen der Aufnahmegeräte. Die Simulation korreliert
multimodale Patientendaten und liefert einen besseren Eindruck der
Gefäßarchitektur und Strömung als die einzelnen Aufnahmeergebnisse.
Es ist möglich die Strömung in den Gefäßen im krankhaften Zustand
darzustellen und die Veränderung der Strömung nach einem operativen
Eingriff vorherzusagen. |
Zur
Anwendung im MediGRID Applikationsportal
fMRI
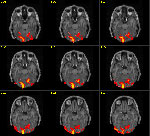 |
Die Demoversion der Anwendung funktionelle Hirnbilddaten basiert
auf den Beispielprogrammen von Prof. Dr. Dr.
Bernarding Stand Juli 2006. Sie soll einen ersten Eindruck
vermitteln wie eine Integration in MediGRID und die Benutzerfühung
im Portal aussehen könnte.
Nach einer manuellen Segmentierung in einer
Bildverarbeitungssoftware (z.B. AMIRA), liegen die Bilder lokal vor
und müssen auf den entsprechenden Rechnerknoten transferiert werden.
Für diese Demo wurde schon ein segmentierter Beispiel-Bilddatensatz
auf alle Gridrechner kopiert. Konvertierungen und Berechnungen der
Simulation erfolgen dann auf einem Gridrechner, danach liegt das
Ergebnis wieder im AVS Format vor und kann zur Visualisierung auf
den lokalen Rechner heruntergeladen werden. |
Zur
Anwendung im MediGRID Applikationsportal
zurück